【讲座笔记】阿斯利康计算机辅助药物设计讲座
目录
利用人工智能加速药物研发——小分子及其他领域 - 总结提纲
前言: 本次在线讲座由BioSolveIT公司主办,邀请了瑞典阿斯利康的Eva Nittinger博士主讲。讲座主题为"利用人工智能加速药物研发——小分子及其他领域"。本文是笔者整理的讲座笔记,供中文研究者参考。
1. 药物发现过程概述
1.1 DMTA循环的核心地位
- DMTA循环:设计(Design) → 制备(Make) → 测试(Test) → 分析(Analyze)
- 时间挑战:从化学起始点(hit)到候选药物(CD)需要3年以上
- 加速目标:如何通过计算方法加速这一过程
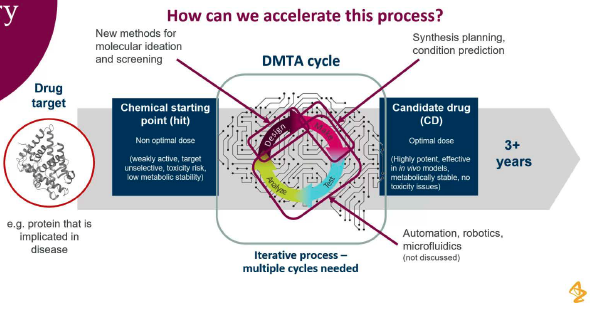
1.2 计算机辅助药物设计的复杂性
- 硅内靶点评估:3D结构预测、结合模式、模式选择
- 化学空间探索:命中识别、化学系列选择、知识产权分析
- 化合物谱优化:多参数(>100)优化、平衡相互关联和对立因素
2. 蛋白质折叠与结构预测
2.1 AlphaFold的成功应用案例
- 分子替换模板:X射线晶体学中的应用
- 功能预测:通过预测数据补充实验数据
- 结构动力学:探索AlphaFold是否能预测蛋白质动态变化
2.2 结构预测方法的局限性
优势领域:
- 单一结构解析
- 强蛋白质-蛋白质相互作用
不适用领域:
- 动态和结构运动
- 新型模式(如PROTACs)
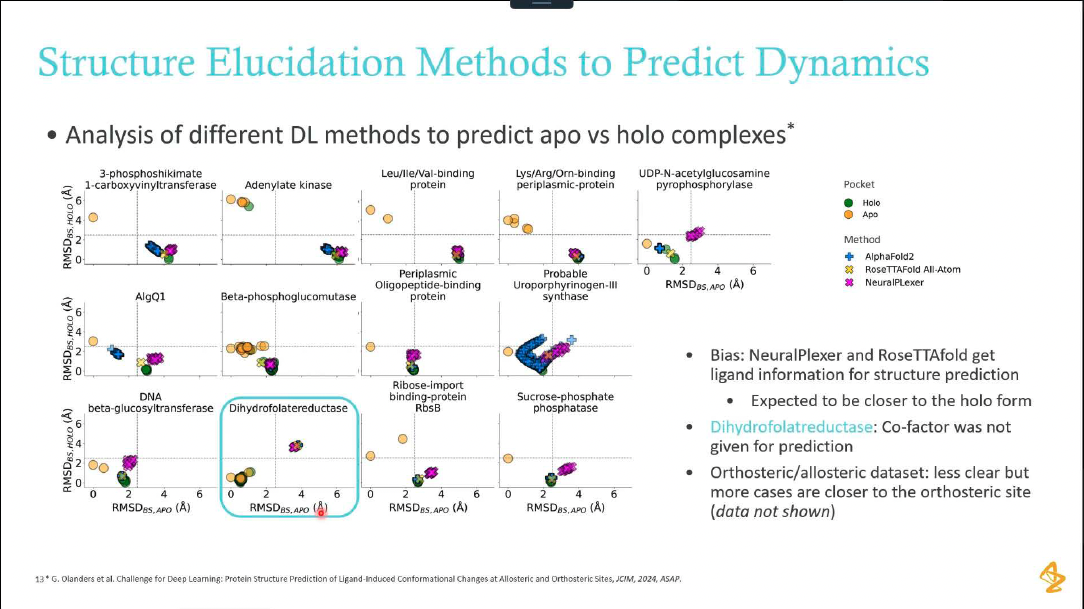
2.3 深度学习方法比较
- 多种方法评估:AlphaFold2、RoseTTAFold、NeuralPlexer等
- 结合状态预测:开放态(apo)vs闭合态(holo)结构预测
- 偏差分析:NeuralPlexer和RoseTTAFold在配体信息获取方面的优势
3. 化学空间探索与利用
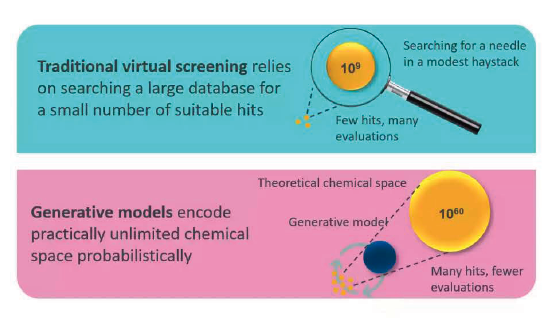
3.1 生成式建模 vs 虚拟筛选
传统虚拟筛选:
- 在大型数据库中搜索,寻找少量合适的命中化合物
- 类似"大海捞针"的过程
生成式模型:
- 以概率方式编码几乎无限的化学空间
- 更高效的化学空间探索
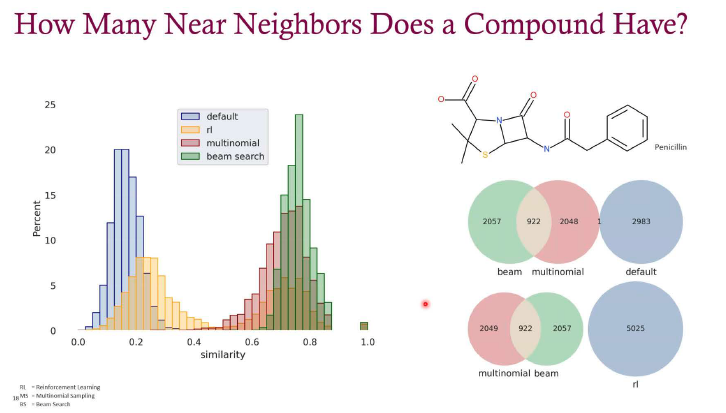
3.2 化合物相似性分析
- 近邻分析:化合物拥有多少个近邻?
- 采样策略对比:默认、强化学习、多项式、集束搜索
- 性能评估:通过REINVENT等方法进行实际应用验证
3.3 化学空间的规模和多样性
- 数据来源:基于26个AZ内部项目的回顾性分析
- 给药途径:口服给药分析
- 关键参数:logD、溶解性、清除率的分析
4. 化合物优化与分子设计
4.1 分子构思与化合物优先级排序
- 挑战:如何过滤和选择?
- 多种策略:
- X射线模型富集对接得分
- 分子动力学探索FEP预测亲和力
- QSAR插值预测活性
4.2 QSAR方法的假设与局限
基本假设:
- 相似性原理:“结构相似的化合物具有相似的活性”
- 线性和加性:化学空间中的线性关系
- 外推前提:从已知数据预测未知数据
主要局限:
- 非加性问题:R²和RMSE在非加性数据上表现显著较差
- 数据依赖性:公共数据(5.1%)和内部数据(9.4%)中存在显著的NA值
- 模型构建重要性:检测非线性对进一步使用至关重要
4.3 深度学习对接 vs 经典方法
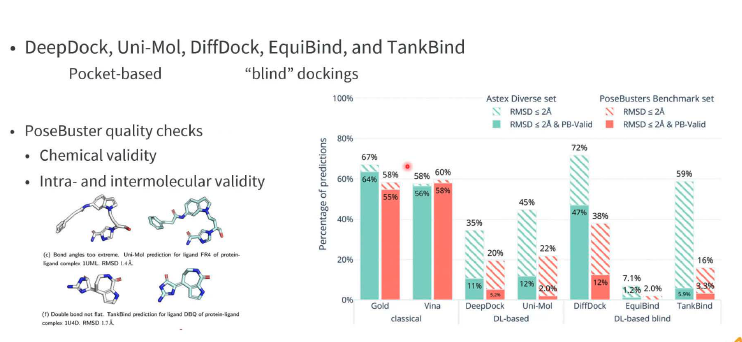
- 方法比较:DeepDock、Uni-Mol、DiffDock、EquiBind、TankBind
- 验证标准:PoseBuster质量检查、化学有效性、分子内和分子间有效性
- 结论:DL方法未能超越经典对接方法
5. 新型模式:PROTAC技术
5.1 PROTAC工作机制
- 三元复合物形成:E3连接酶 + PROTAC分子 + 目标蛋白(POI)
- 降解过程:POI泛素化 → 蛋白酶体降解
- 优势:针对"不可药化"靶点的新策略
5.2 PROTAC三元复合物预测
研究设置:
- 数据集:9个PROTAC三元复合物
- 序列选择:晶体结构(~130氨基酸) vs 全长序列(1647氨基酸)
- 预测挑战:基于E3和POI预测,无PROTAC分子本身信息
预测结果分析:
- 全长序列:E3和POI之间无预测接触
- 晶体序列:正确预测个别蛋白质,但E3和POI相对位置不正确
- 人工连接:降低结构变异,但相对/旋转定位仍不正确
5.3 结构预测方法的进一步结论
不适用场景:
- 动态和结构运动
- 新型模式(如PROTACs)
- 需要辅助折叠改善预测
改进方向:
- AF2 → AF3:某些改善但不一致
- AF3^multimer → AF3^16:多数情况下有改善
6. 降解效率建模
6.1 数据集构建
- PROTAC-DB数据:包含独特SMILES、独特靶点、共享SMILES的综合数据库
- 数据处理流程:正则表达式 → 合并 → 标准化细胞系和E3配体 → 应用过滤器和定义
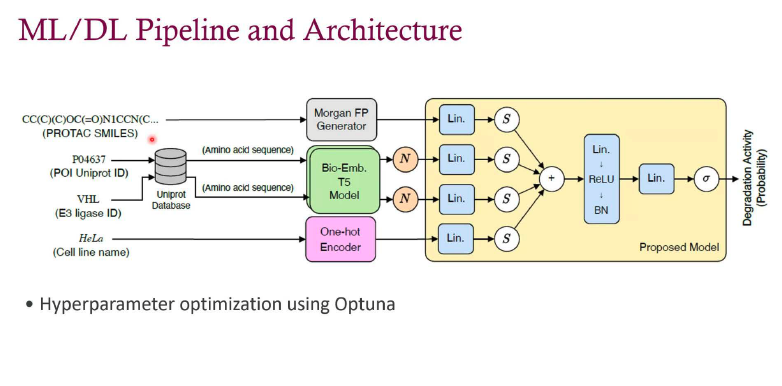
6.2 机器学习模型架构
- 输入特征:PROTAC SMILES、POI UniProt ID、E3配体ID、细胞系名称
- 模型组件:Morgan FP生成器、Bio-Emb T5模型、One-hot编码器
- 超参数优化:使用Optuna进行优化
6.3 模型性能评估
- 数据拆分策略:标准分割、相似性分割、靶点分割
- 评估指标:验证准确率、测试准确率、ROC AUC
- 消融研究:确定各架构组件的重要性
7. 未来发展方向
7.1 技术挑战
- 生成准确的三元结构:仍是开放性挑战
- 辅助折叠潜力:是否能改善预测效果?
- PROTACsplitter开发:为PROTAC降解预测提供信息
7.2 实际应用考量
现实问题:
- 生成模型优势:在新化合物发现方面表现良好
- 高通量对接限制:结构柔性仍是问题
- 筛选过程挑战:需要正确识别和预测化合物性质
数据和模型改进:
- 更好的模型和更好的数据是关键
- 非加性作为预测目标:识别和预测非加性行为
- 案例依赖性:目前仍然高度依赖具体案例,缺乏标准化解决方案
8. 总结:炒作还是现实?
8.1 技术现状评估
- 生成模型:在新化合物发现方面确实有效
- 结构预测:高通量对接和结构柔性仍有问题
- 实际应用:筛选过程需要正确的化合物性质识别和预测
8.2 关键成功因素
- 数据质量:更好的数据是基础
- 模型改进:持续的算法和方法学创新
- 非加性预测:作为理想的预测和识别事件
- 标准化需求:目前仍然高度依赖案例,缺乏通用解决方案
核心结论:虽然AI在药物发现领域展现出巨大潜力,但当前技术仍面临显著挑战。成功的关键在于结合更好的数据、改进的模型以及对方法局限性的清晰认识。特别是在PROTAC等新兴治疗模式方面,仍需要大量的技术创新和方法学突破。
